2023-11-06,Galaxy生信云平台 UseGalaxy.cn 新增circos圈图绘制工具。
Graph/Display Data
- Circos visualizes data in a circular layout (Galaxy Version 0.69.8+galaxy12)
Circos(Krzywinski等人,2009年)是一个用于以圆形布局可视化数据的软件包。这使得Circos非常适合探索对象或位置之间的关系。Circos图表已经出现在成千上万的科学出版物中。尽管最初设计用于可视化基因组数据,但它可以根据任何领域的数据创建图表。
文章图形复现
图形/数据来源:
Teles Alves I, Hiltemann S, Hartjes T, et al. Gene fusions by chromothripsis of chromosome 5q in the VCaP prostate cancer cell line. Hum Genet. 2013;132(6):709-713. doi:10.1007/s00439-013-1308-1
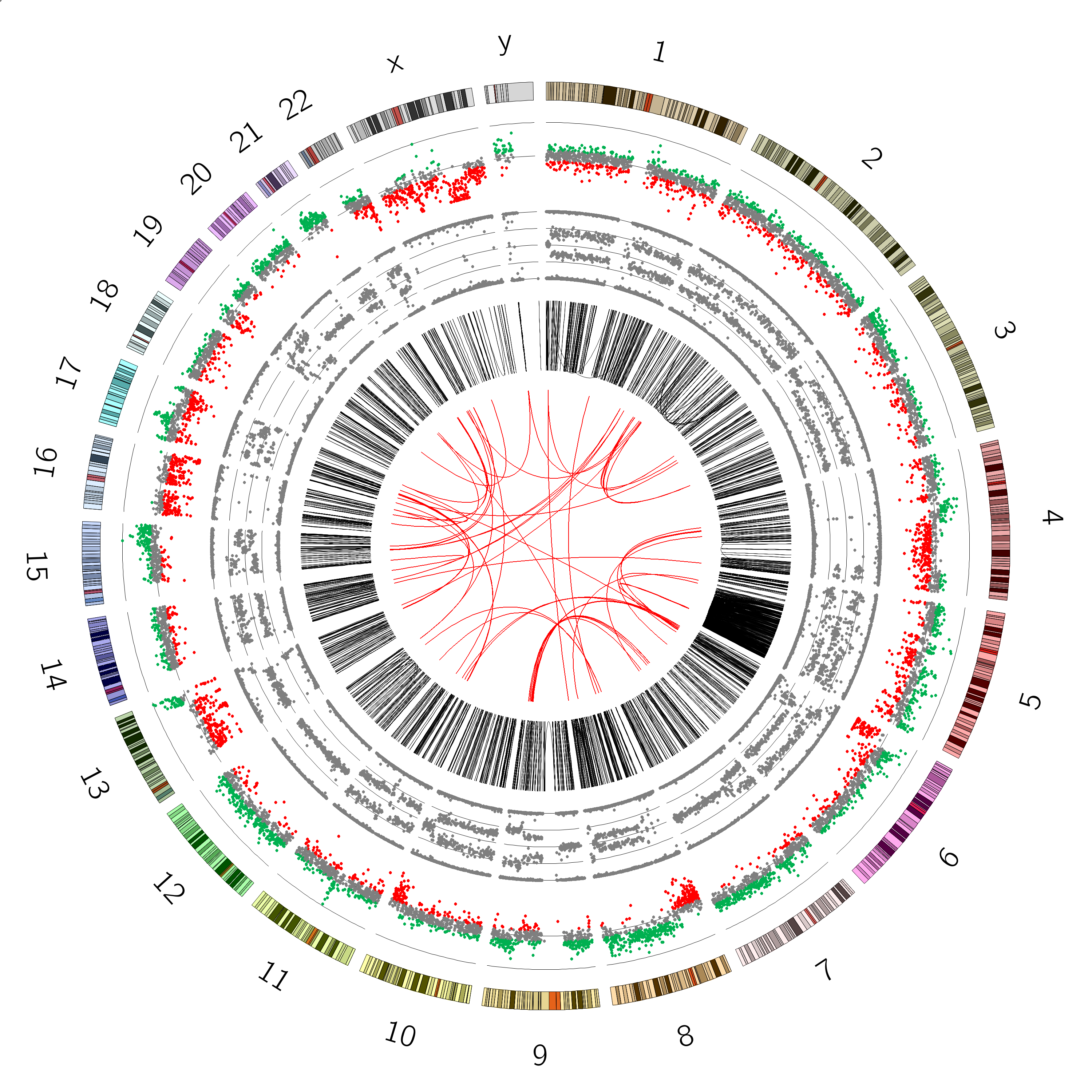
在这篇文章中,各种来源的数据被整合到一个综合的Circos图中。下面我们就来复现这个图,首先我们先了解一下Circos的基础。
1. Circos基础
1.1 外层骨架(Ideogram)
对于基因组数据,通常是染色体,但也可能是物种、基因,或者是另一个分辨率级别,这取决于你想要展示的关系。对于非基因组数据,这可能是人群中的个体、国家,或者是你想用来进行分组的数据的任何其他主要方面。
1.2 数据轨道(Data Tracks)
在骨架之内,我们可以绘制数据轨道。有不同类型的图表可用,例如:
- 散点图(Scatter plot)
- 线图(Line plot)
- 直方图(Histogram)
- 热图(Heatmap)
- 方块图(Tile)
- 文本(Text)
- 连接(Link)
- 条带(Ribbons)
轨道可以进行很多定制,一些相关的概念包括:
-
Radius:轨道的半径决定了它在中心(0)和表意图(1)之间的位置。
-
Rules:可以定义规则,根据数据点的值改变数据点的颜色,例如。
-
Axes and backgrounds:可以在数据轨道上绘制轴和背景。
2. 复现步骤
工具位置:UseGalaxy.cn首页 > Graph/Display Data > Circos visualizes data in a circular layout
或者:https://usegalaxy.cn/root?tool_id=toolshed.g2.bx.psu.edu/repos/iuc/circos/circos/0.69.8+galaxy12
2.1 数据准备
可以下载原文中的数据上传到网站:
https://zenodo.org/record/4494146/files/VCaP_Copy-Number.tsv
https://zenodo.org/record/4494146/files/VCaP_B-allele-Frequency.tsv
https://zenodo.org/record/4494146/files/VCaP-highConfidenceJunctions.tsv
https://zenodo.org/record/4494146/files/hg18_karyotype_withbands.txt
或者用网站上整理好的共享数据:
网站首页(UseGalaxy.cn)> 数据共享 > 数据库 > circos,该目录下有4个测试文件:
- hg18_karyotype_withbands.txt,染色体骨架文件
- circos-sv.tsv,结构变异信息
- circos-cnv.tsv,拷贝数变异信息
- circos-baf.tsv,次等位基因频率
2.2 Ideogram
由于圈图要呈现的数据很多,参数设置复杂,因此不可能一次就画好,而是有一个不断迭代的过程,一圈一圈地画。
操作:设置Ideogram
我们首先只设置骨架文件,其他参数保持默认:
运行看看结果:
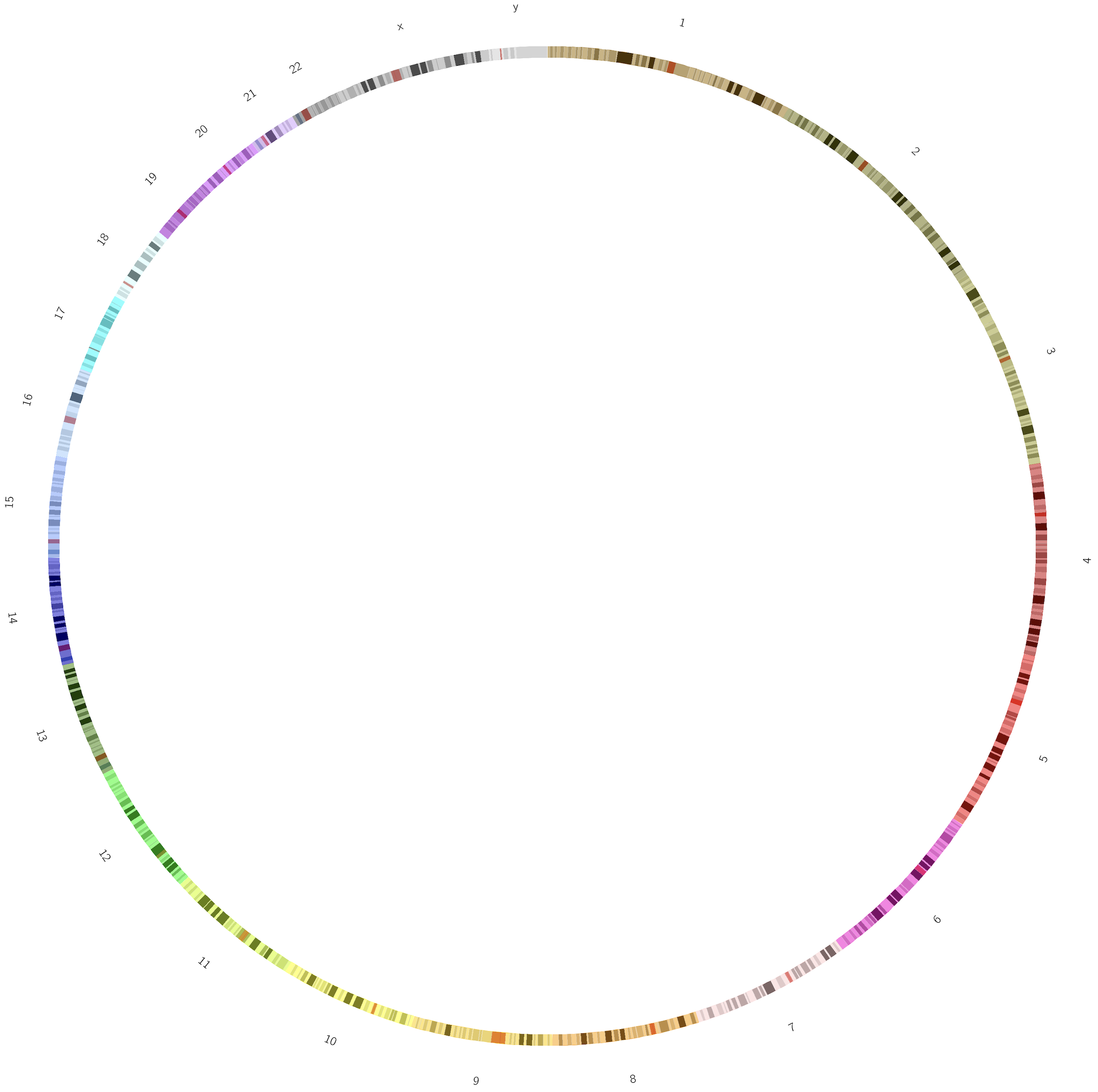
可以看到,只有一个圈。
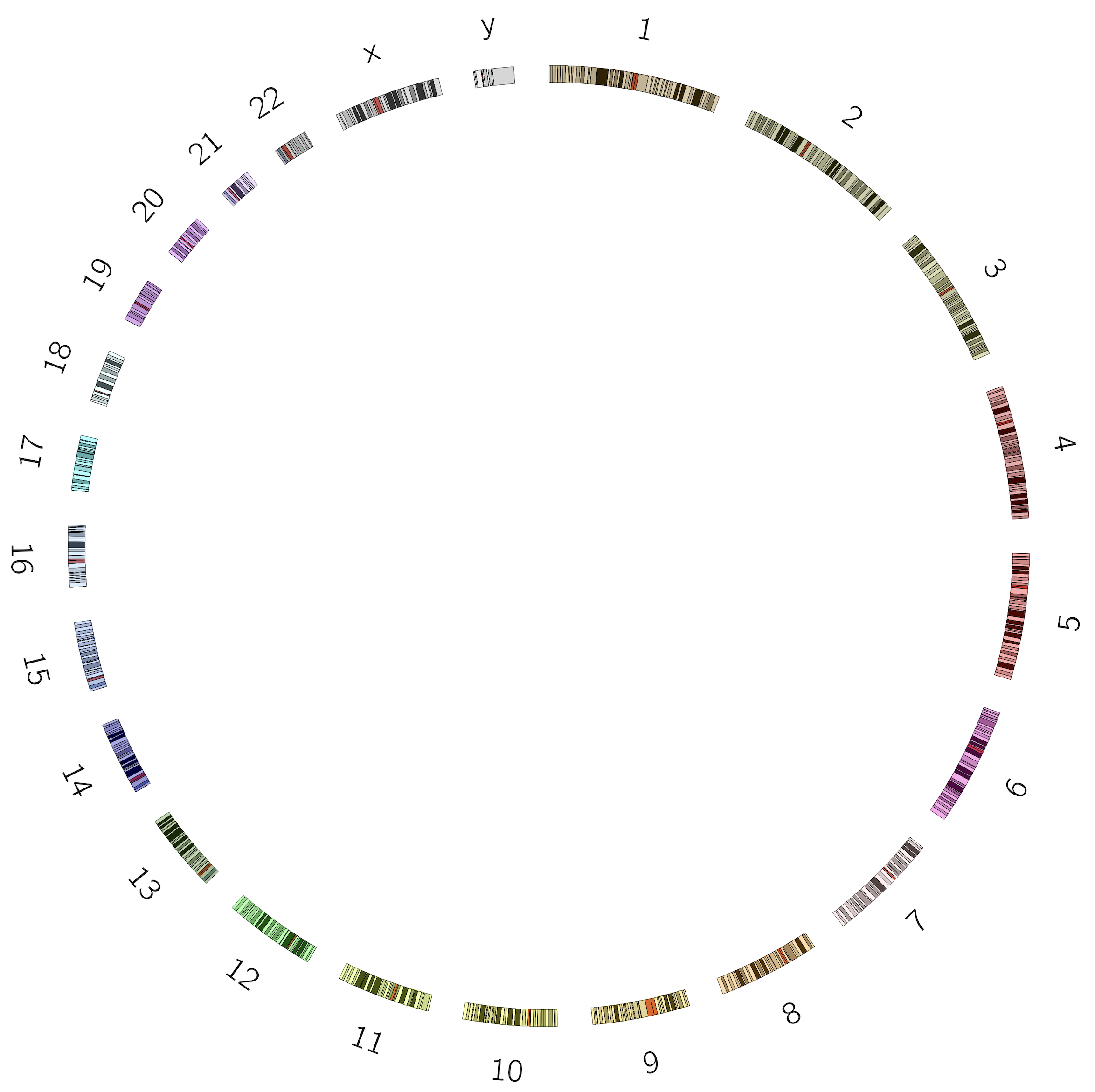
我们接着设置:

点击“Run Tool”,等待运行结束,点击结果文件右边的铅笔图标,将文件改名为:Circos Plot ideogram。
小知识:
染色体定义的格式如下:chr - ID LABEL START END COLOR
前两个字段始终是chr,表示该行定义了一个染色体,以及 -。第二个字段定义了父结构,仅用于染色体条带定义。
ID 是数据文件中使用的标识符,而 LABEL 是将出现在图像上的骨架旁边的文本。
START和END定义了染色体的大小。核型文件应该存储整个染色体的大小,而不仅仅是您希望绘制的区域。我们可以使用其他参数仅绘制数据的子集(例如,只有一个染色体)。
COLOR参数是可选的,要使用内置的颜色方案,请再次在此列中使用chr1、chr2等。
2.3 数据轨道一:结构变异
结构变异需要提供两个断点的位置,如下:
chromosome start end chromosome start end
- 第一个断点的染色体编号,开始位置,结束位置
- 第二个断点的染色体编号,开始位置,结束位置
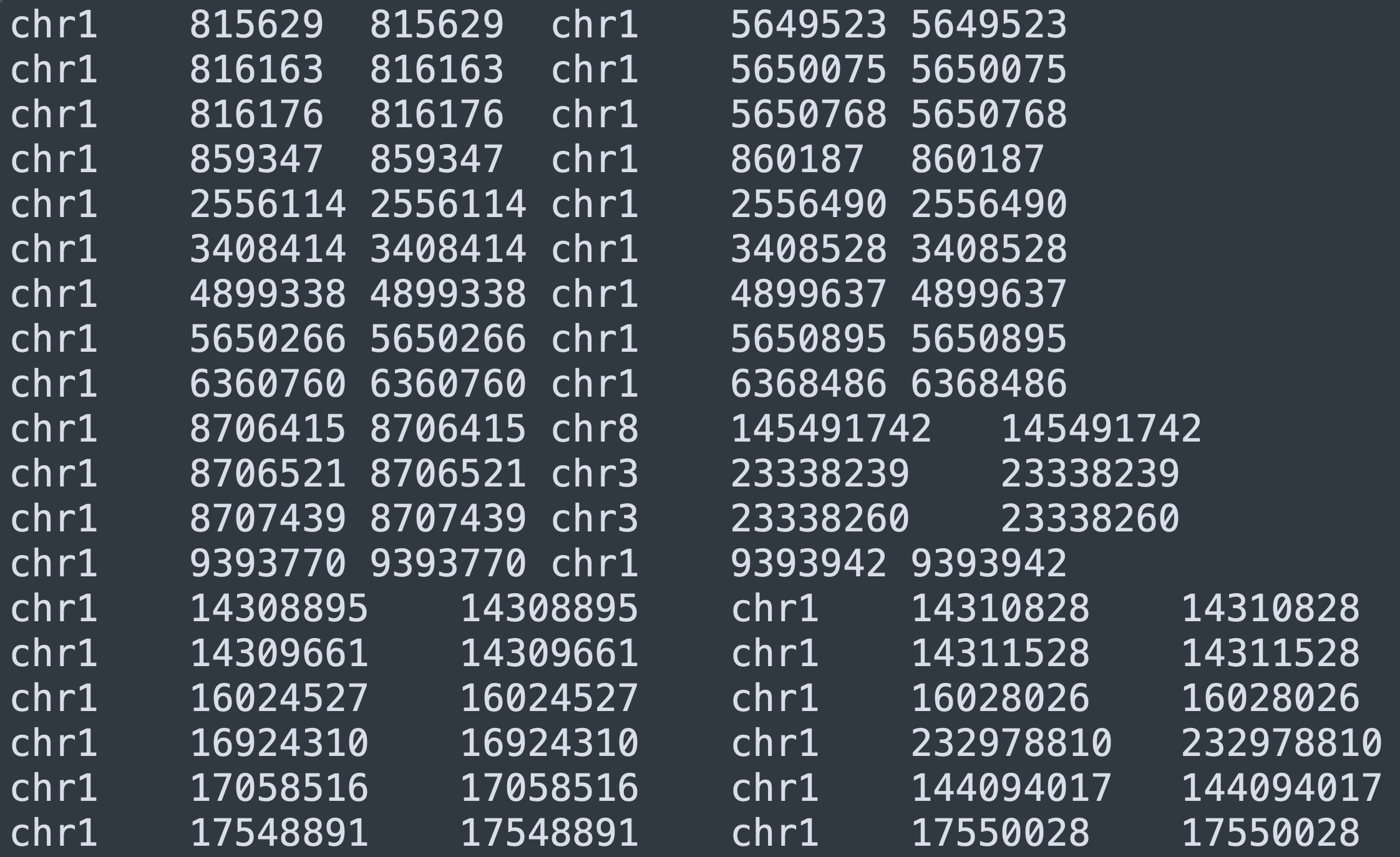
操作:添加结构变异的连线轨道
-
点击右侧历史记录中的Circos Plot ideogram文件名,点击“Run Job Again”(一个转圈的箭头)
-
结构变异轨道参数设置:

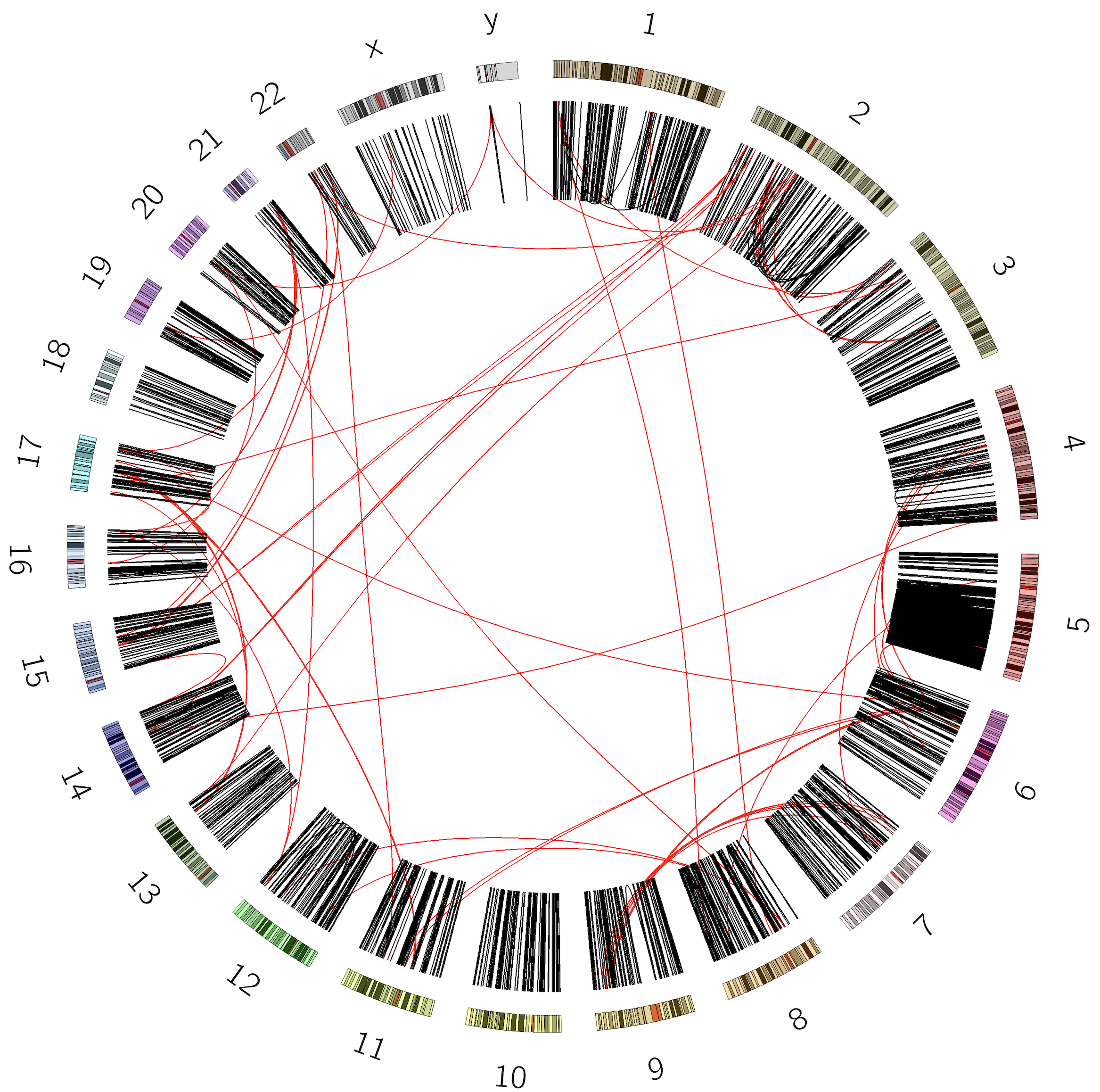
可以看到,5号染色体有许多染色体内的结构变异,我们可以单独画出5号染色体的结构变异。
最后, 将结果文件改名为:Circos Plot SVs
操作:只画5号染色体
-
重新运行刚才的工具
-
改变参数设置:

得到如下图片:

2.4 数据轨道二:拷贝数变异
拷贝数变异需要准备的数据格式:
chr start end value
- chr,染色体
- chr,变异开始位置
- end, 变异结束位置
- value,变异倍数
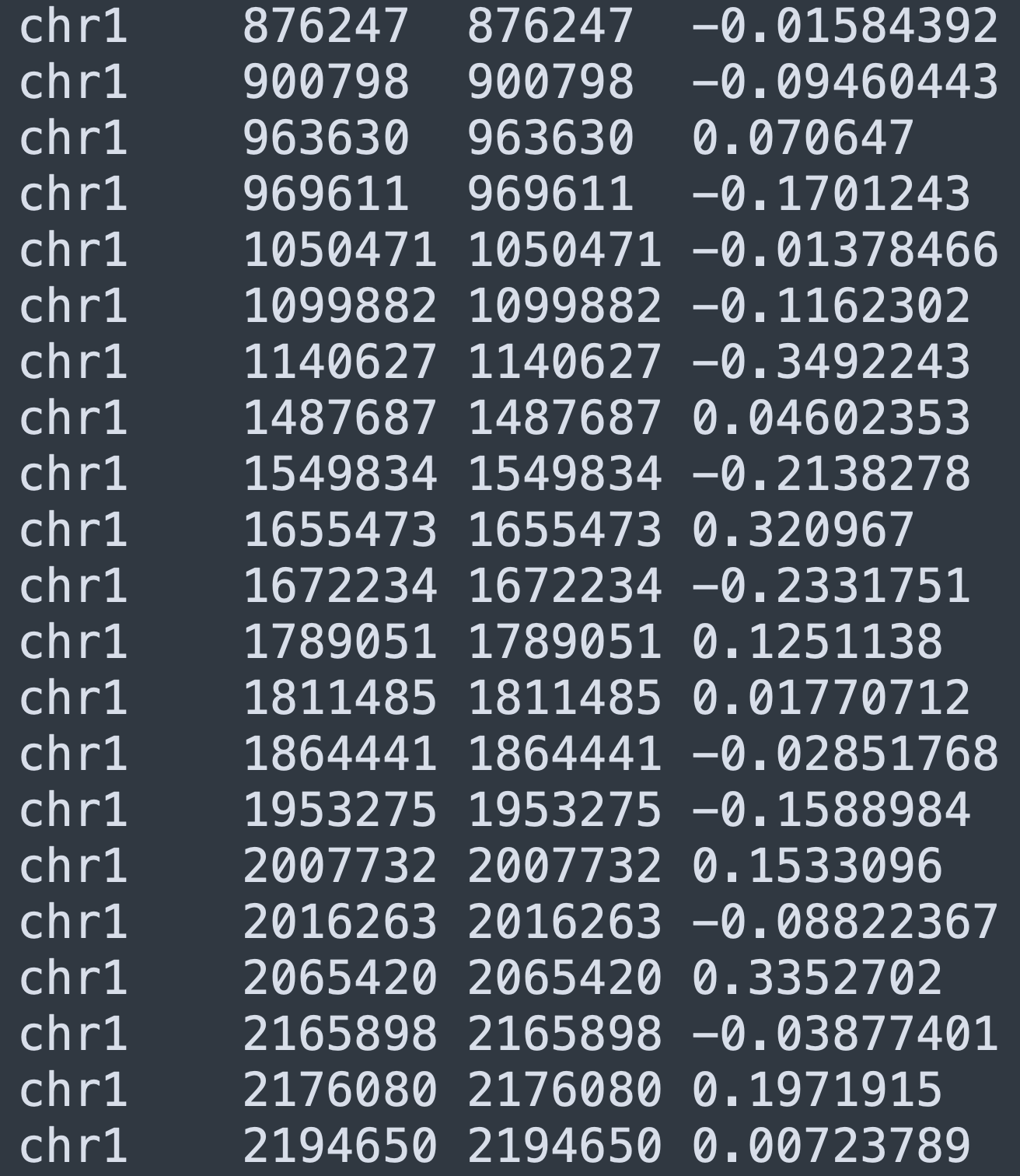
操作:增加拷贝数轨道
-
重新运行画“Circos Plot SVs”的工具。
-
增加一个散点图轨道到图形中:

结果如下:
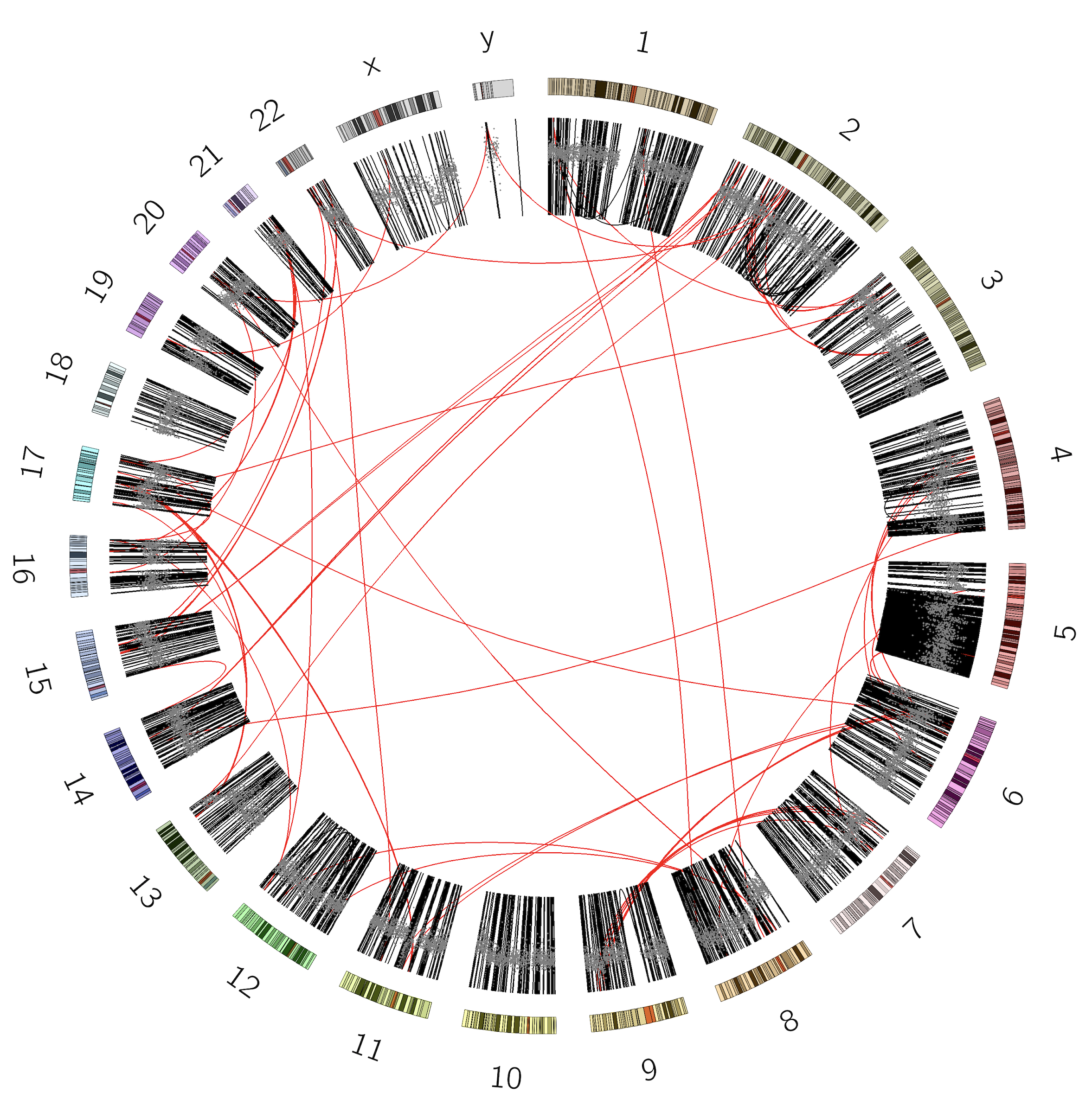
可以看到,拷贝数轨道与结构变异轨道重叠了。这是因为没有个性结构变异轨道的半径设置:
- 重新运行工具,改变以下参数:
In "Link Tracks":
- In "1: Link Data":
- "Inside Radius": 0.75
应该看到这样的图:
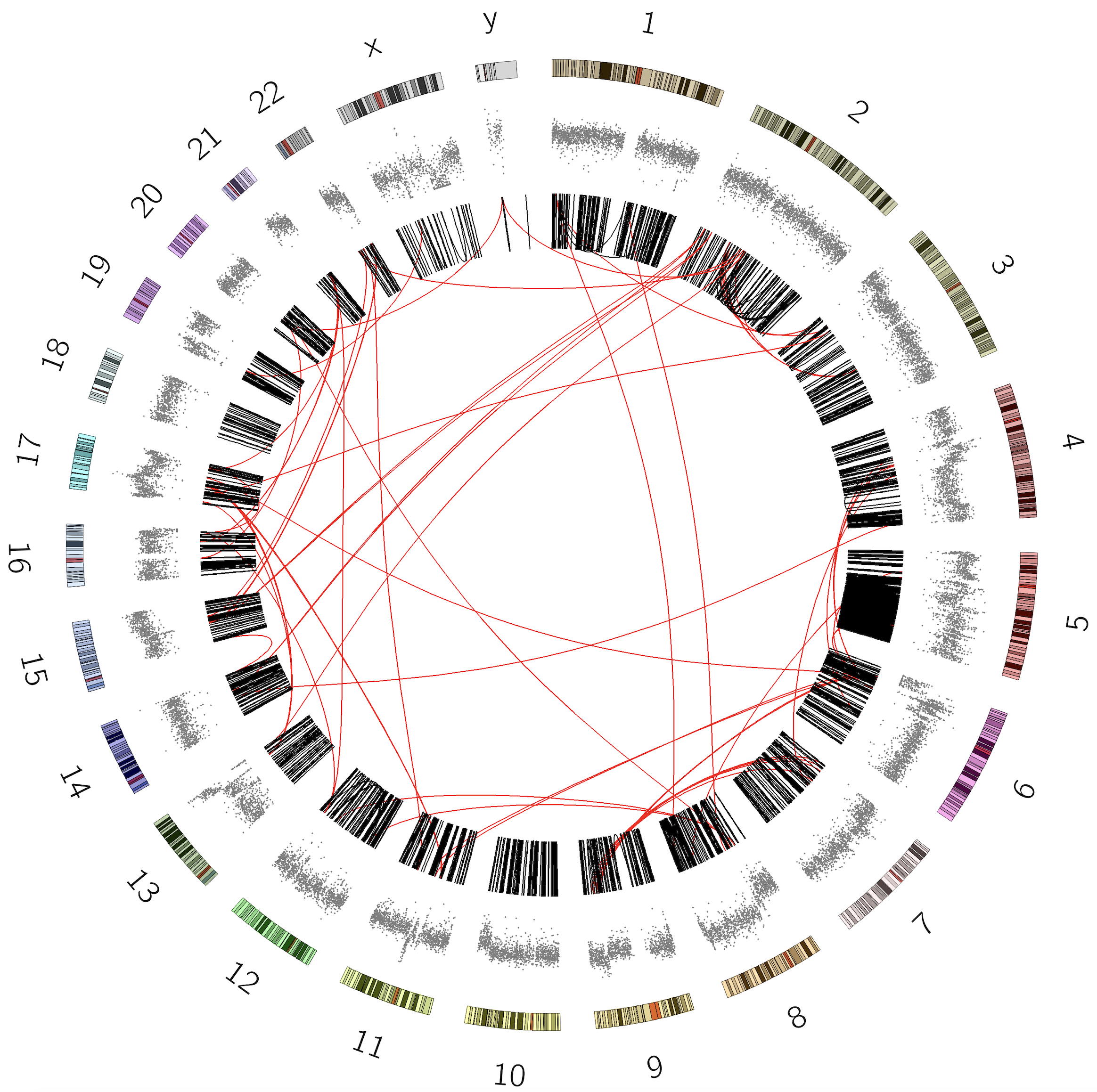
操作:用不同颜色标记拷贝数变异状态
-
重新运行上一步的结果
-
在2D data track的拷贝数轨道中,增加以下规则:

应该得到如下结果:
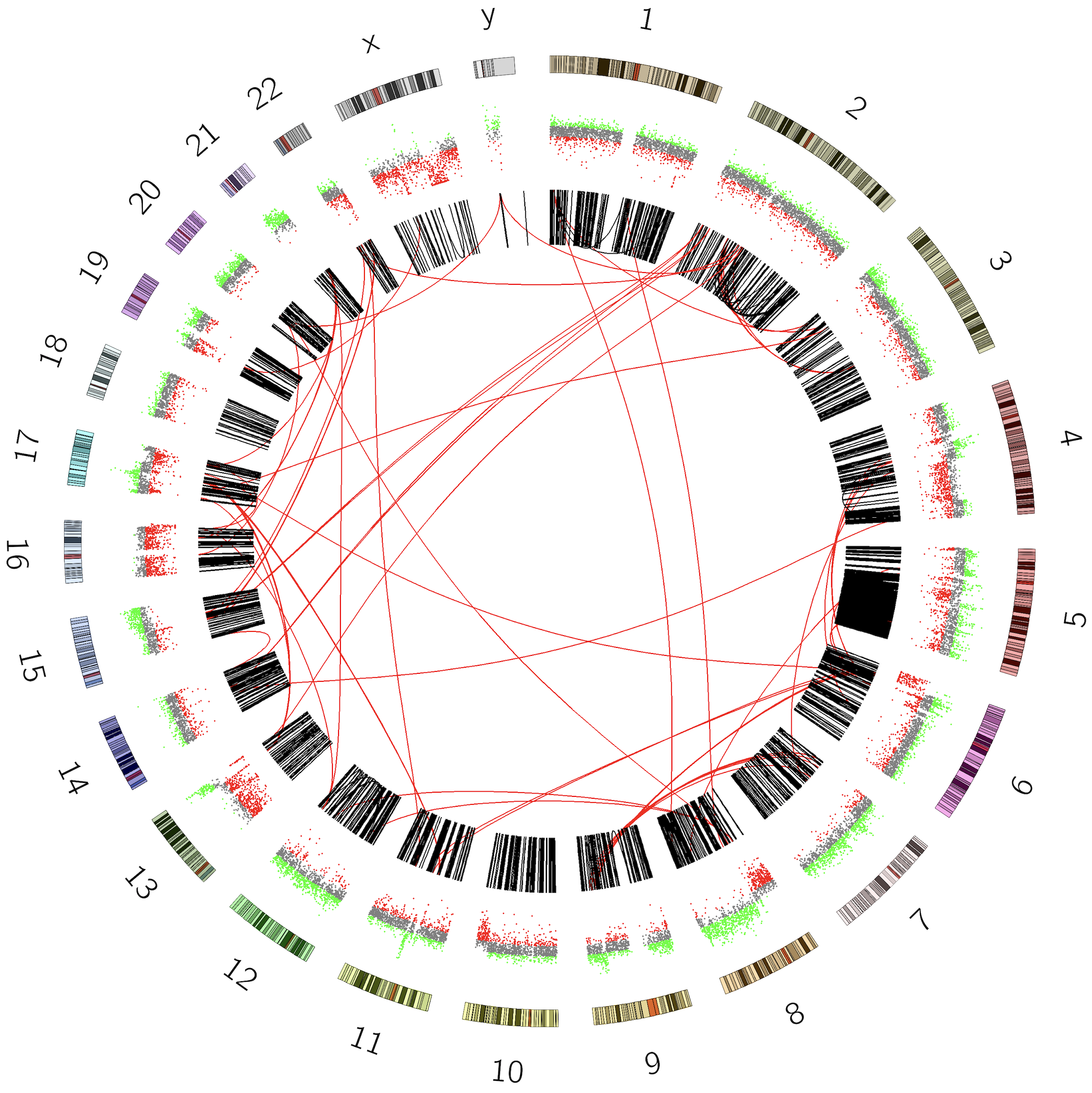
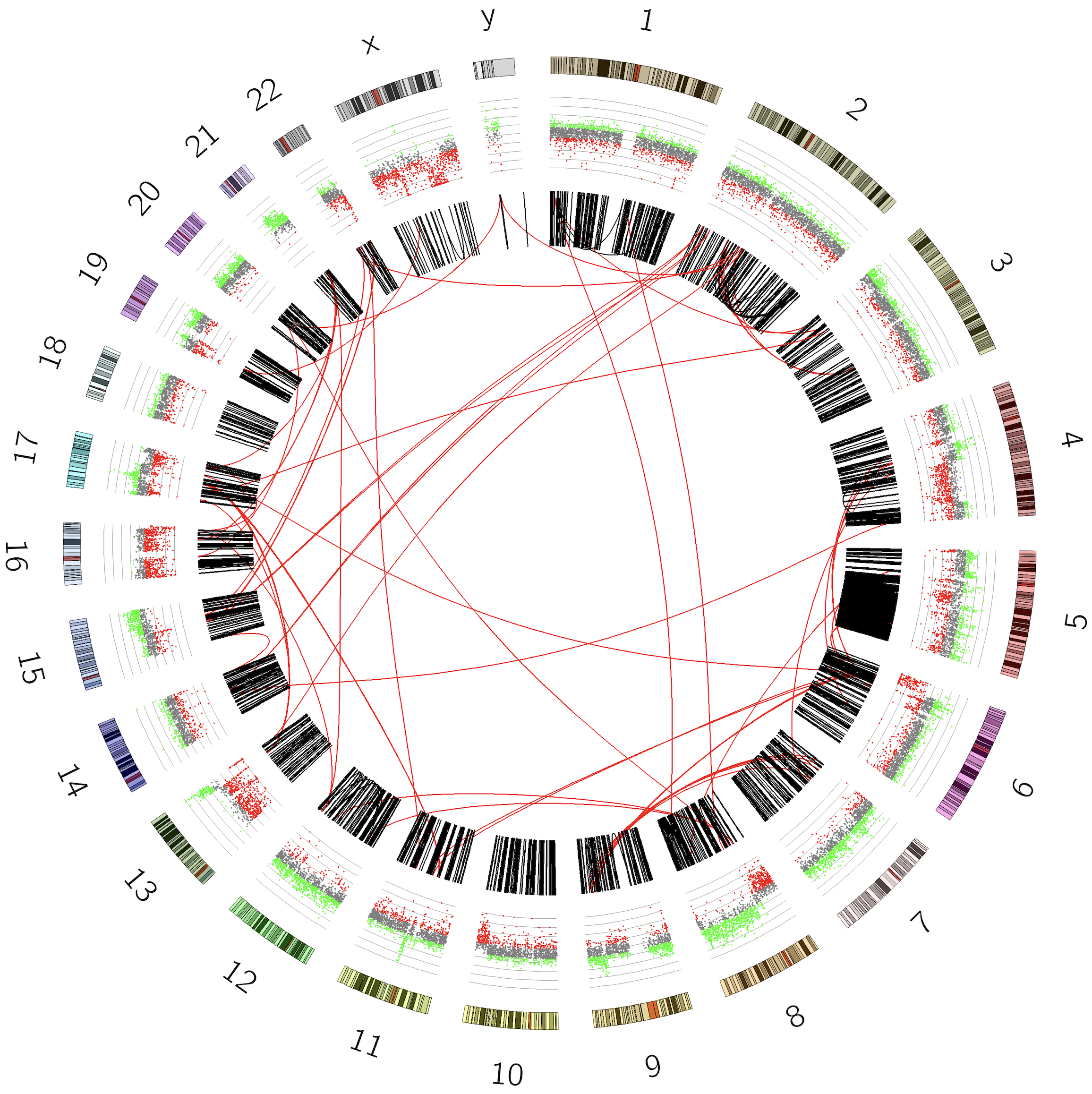
操作:为拷贝数轨道增加坐标轴
-
重新运行刚才的结果
-
在2D data track中的拷贝数轨道设置页面:

结果如下:
最后, 重命名结果文件为:Circos Plot CopyNumber
2.5 数据轨道三:次等位基因频率
数据格式与拷贝数变异的类似,也是准备4列数据:
chr start end value
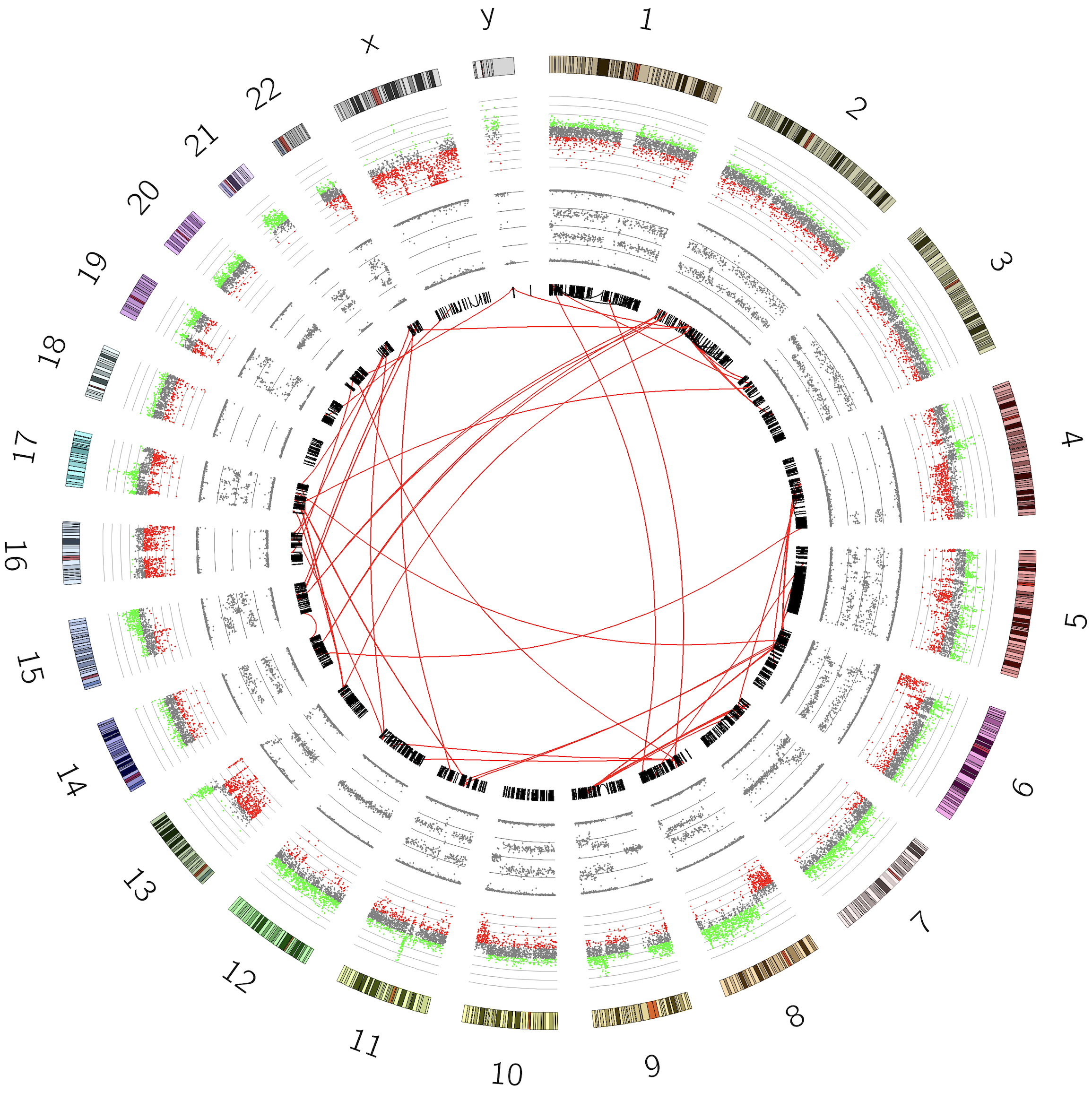
操作:增加次等位基因频率到图中
-
重新运行刚才的工具(Circos Plot CopyNumber)
-
增加新的散点图轨道到图中:

最后, 重命名结果文件:Circos Plot BAF
结果图:
2.6 存在问题:如何将结构变异轨道拆分成两个
操作:两个连接轨道
-
重新运行刚才的工具(Circos Plot BAF)
-
配置两个单独的连接轨道


最后结果:
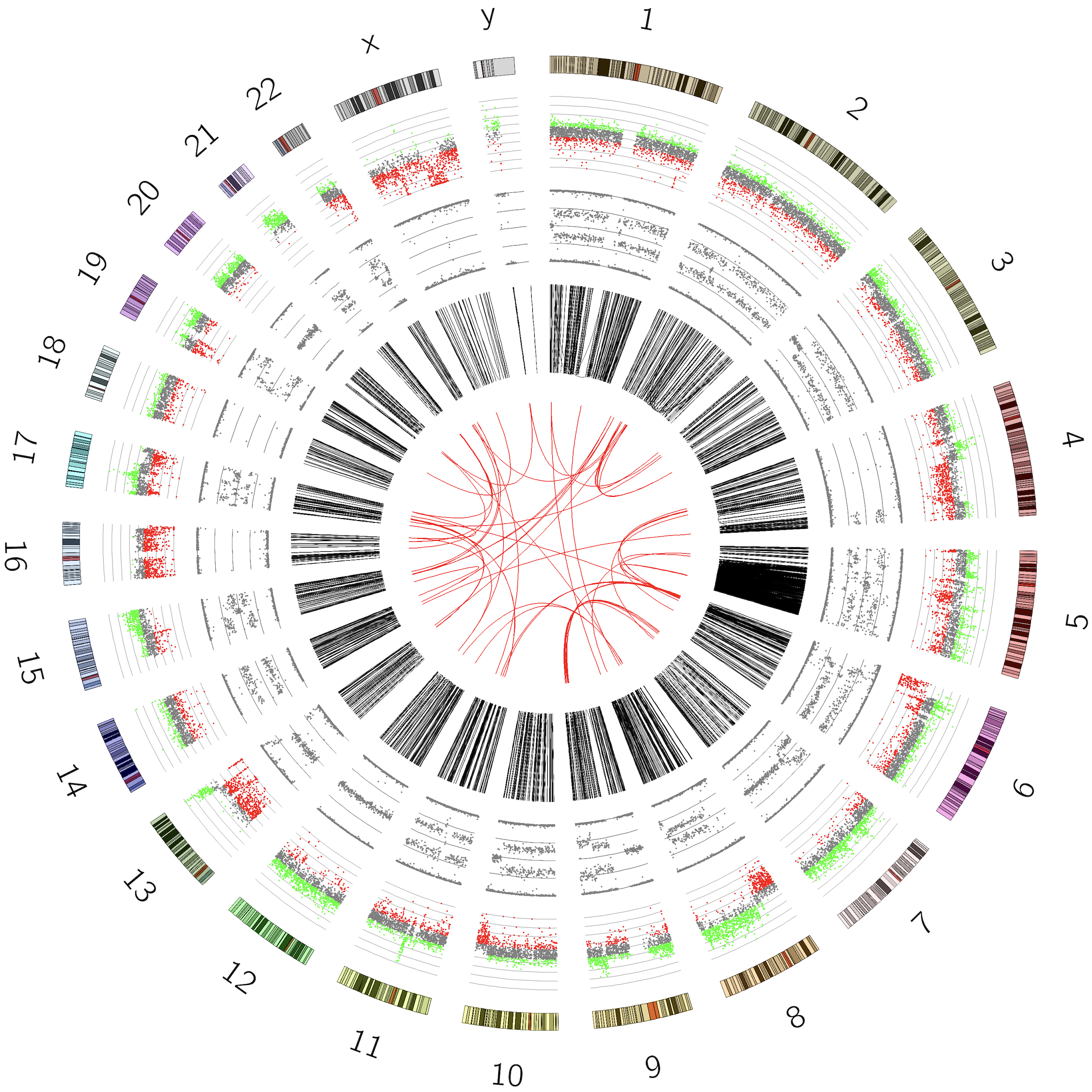
可以看到,跟原图完全一样,可以说复现效果达到了100%。
友情提示
使用问题请加入Galaxy中国交流群: 925694514(QQ群,免费加入),或者关注公众号:简说基因。

