文本文件是生物信息学中应用非常广泛的文本格式,甚至可以说是最重要的文件格式,比如常见的测序下机数据Fastq、参考基因组保存格式Fasta、比对文件SAM,以及突变列表VCF,它们都是文本文件。熟练地进行文本文件的处理,对于生信数据分析来说非常重要。比如为特定程序准备相应的输入文件,或者从结果文件中提取需要的信息。
文本文件的操作贯穿生信数据处理的始终,甚至有人开玩笑说,搞生信分析,就是进行各种文件格式的转换。有时候确实可以这么说,因为现在有许多软件/包都写得非常完善了,只需要准备好相应的输入文件,一行代码即可完成分析。
可以说,善于文本文件的处理,生信数据分析就站在了一个非常高的起点。UseGalaxy.cn平台有非常强大的文本数据处理功能,本文就来系统地介绍这些文本处理工具的用法。
测序数据
- Libraries > Table data: heatmap_test.tsv,一个模拟的 RNA-seq 表达量矩阵数据。
1. 文本操纵(Text Manipulation)
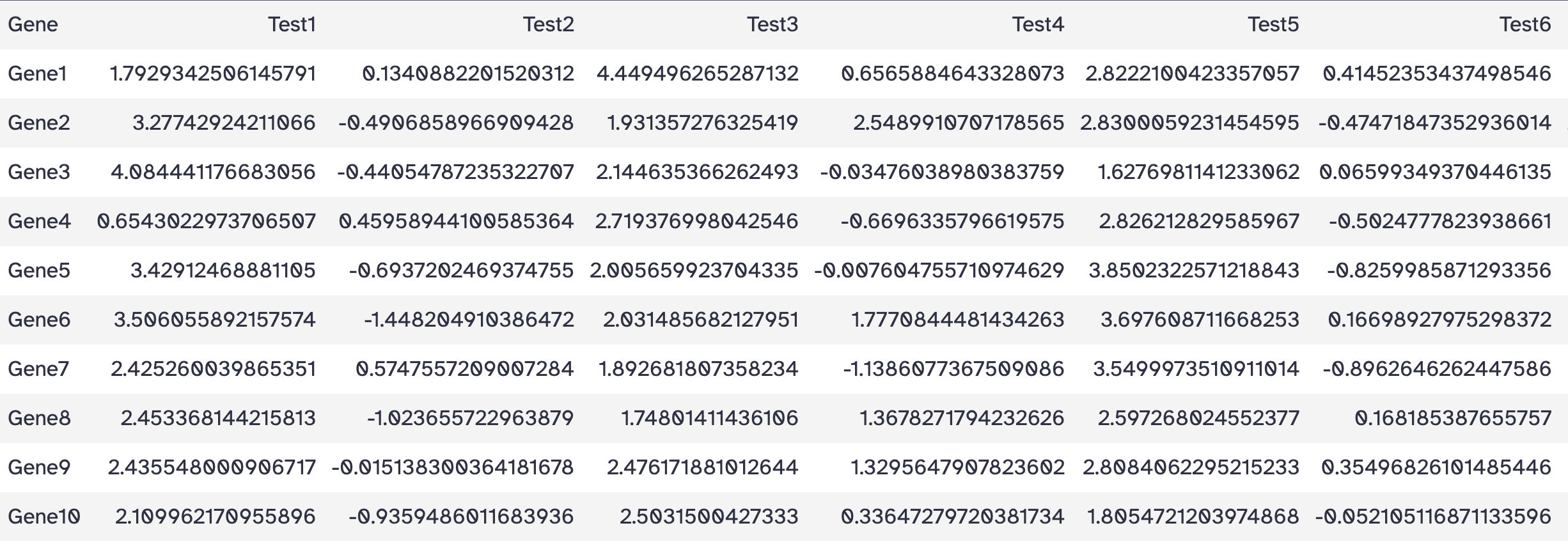
选择开始的多少行
工具:Text Manipulation > Select first lines from a dataset
目标:选择前10行
操作:
- Select first: 11 (因为文件有表头,所以这里设置为前11行)
- from: heatmap_test.tsv
结果:
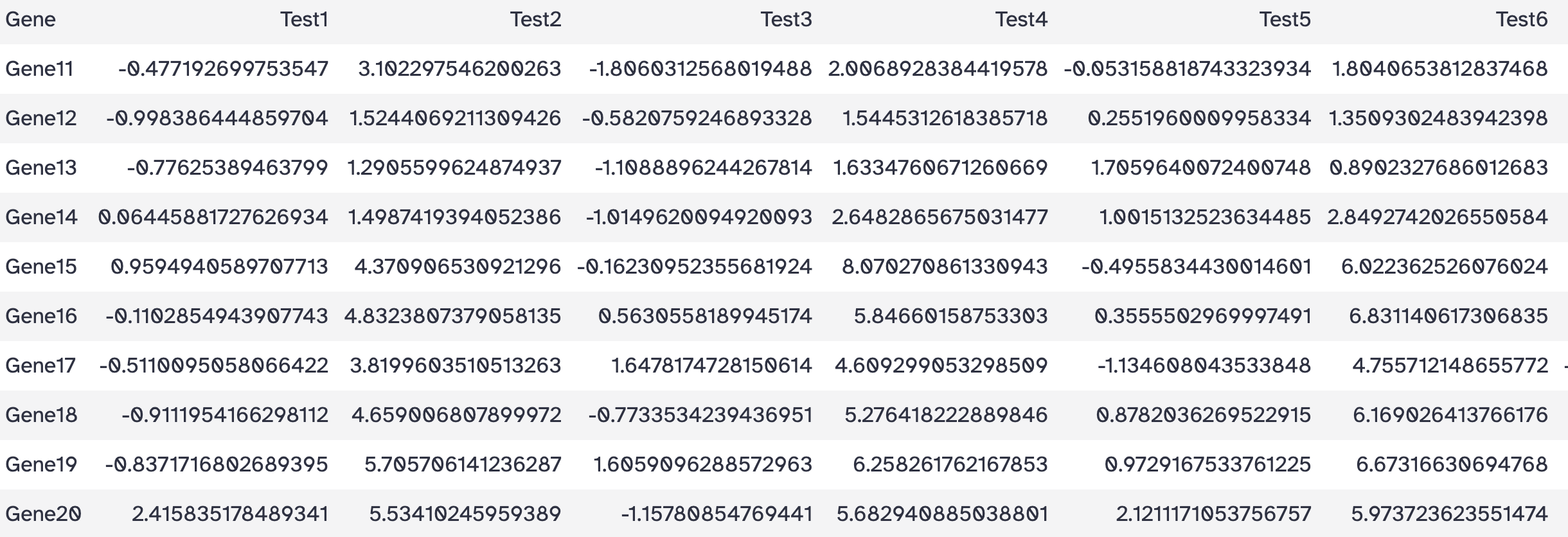
选择结尾的多少行
工具: Text Manipulation > Select last lines from a dataset
目标: 选择结尾的10行
操作:
- Select last: 10
- from: heatmap_test.tsv
- Dataset has a header: Yes
结果:
随机选择多少行
工具: Text Manipulation > Select random lines from a file
目标: 随机选择5行
操作:
- Randomly select: 5
- from: heatmap_test.tsv
结果:
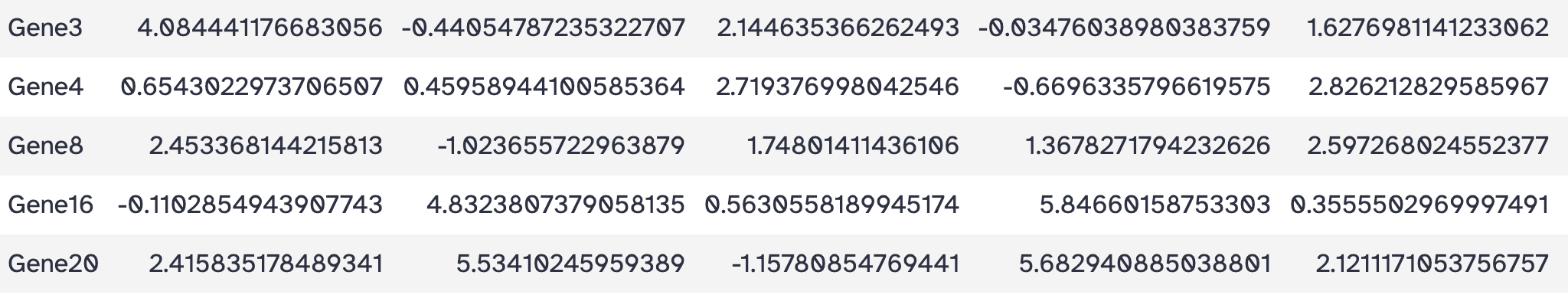
删除开始的多少行
工具: Text Manipulation > Remove beginning of a file
目标: 删除表头
操作:
- Remove first: 1
- from: heatmap_test.tsv
结果:
选择列
工具: Text Manipulation > Cut columns from a table
目标: 选择第1,2,5列
操作:
- Cut columns: c1,c2,c5
- from: heatmap_test.tsv
结果:
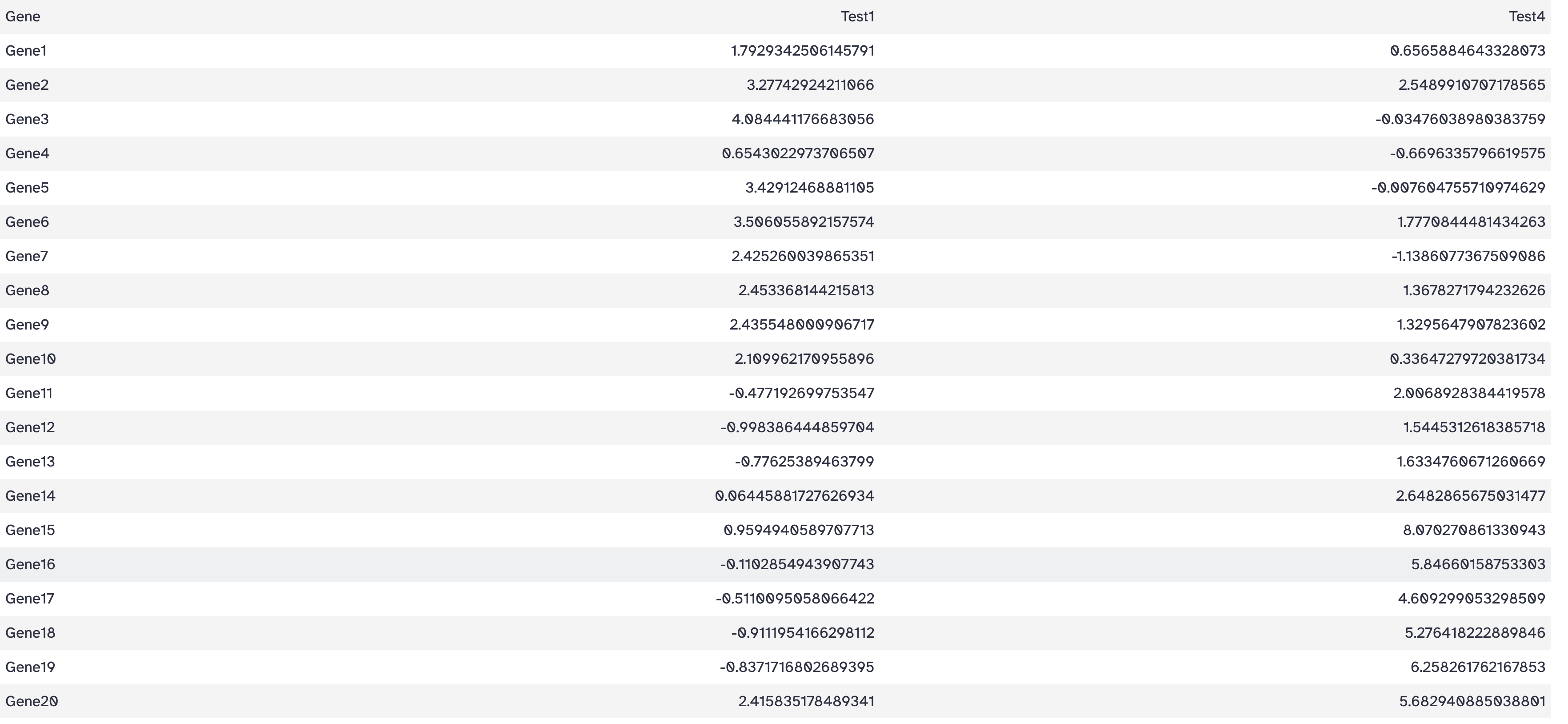
增加列到一个数据表
工具: Text Manipulation > Add column to an existing dataset
目标: 在最后一列增加一个+号
操作:
- Add this value:+
- to Dataset: Remove beginning on data 1
- Iterate?: NO
结果:
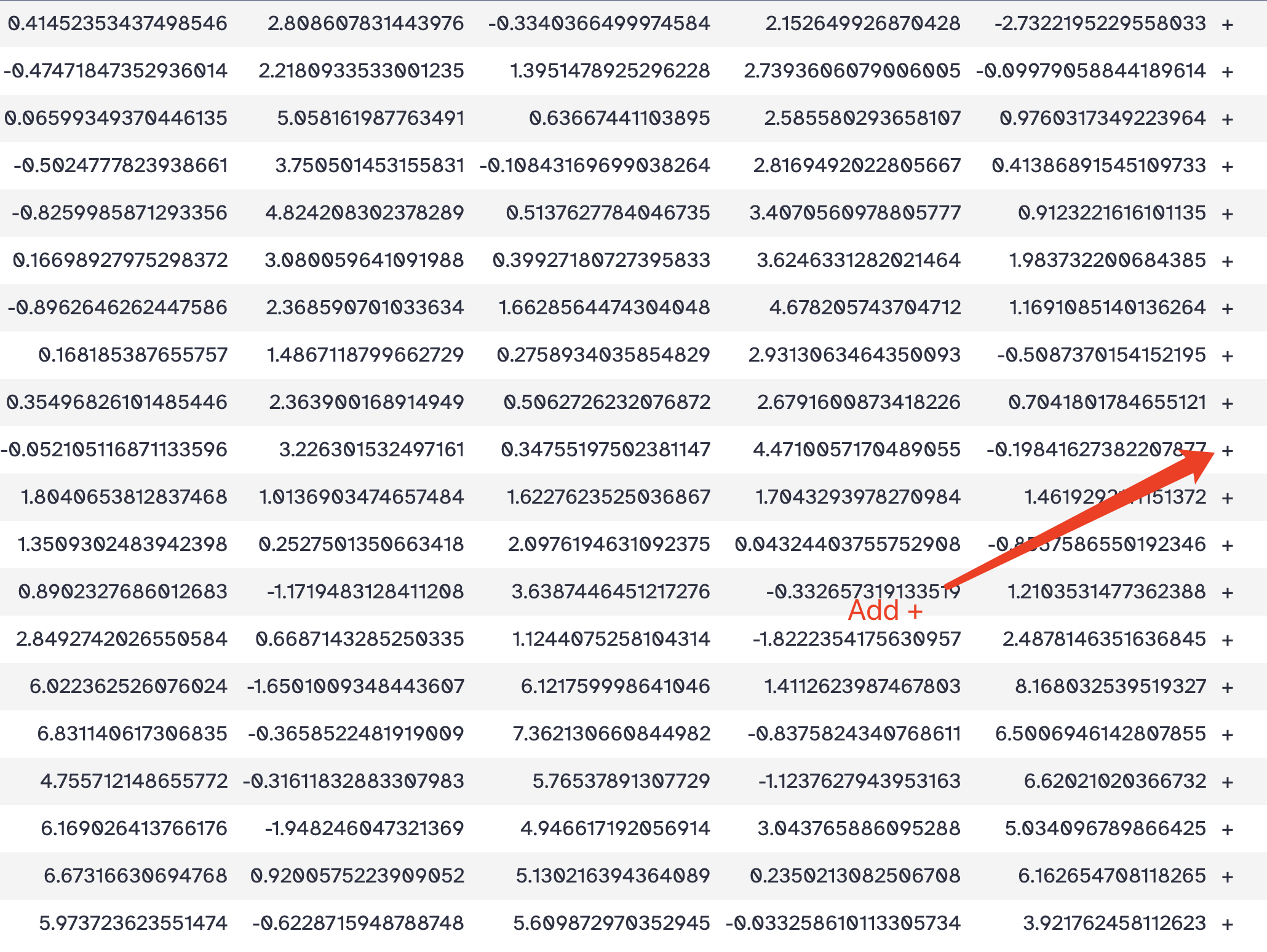
目标: 在最后一列增加从1开始不断迭代的编号
操作:
- Add this value:1
- to Dataset: Remove beginning on data 1
- Iterate?: YES
结果:

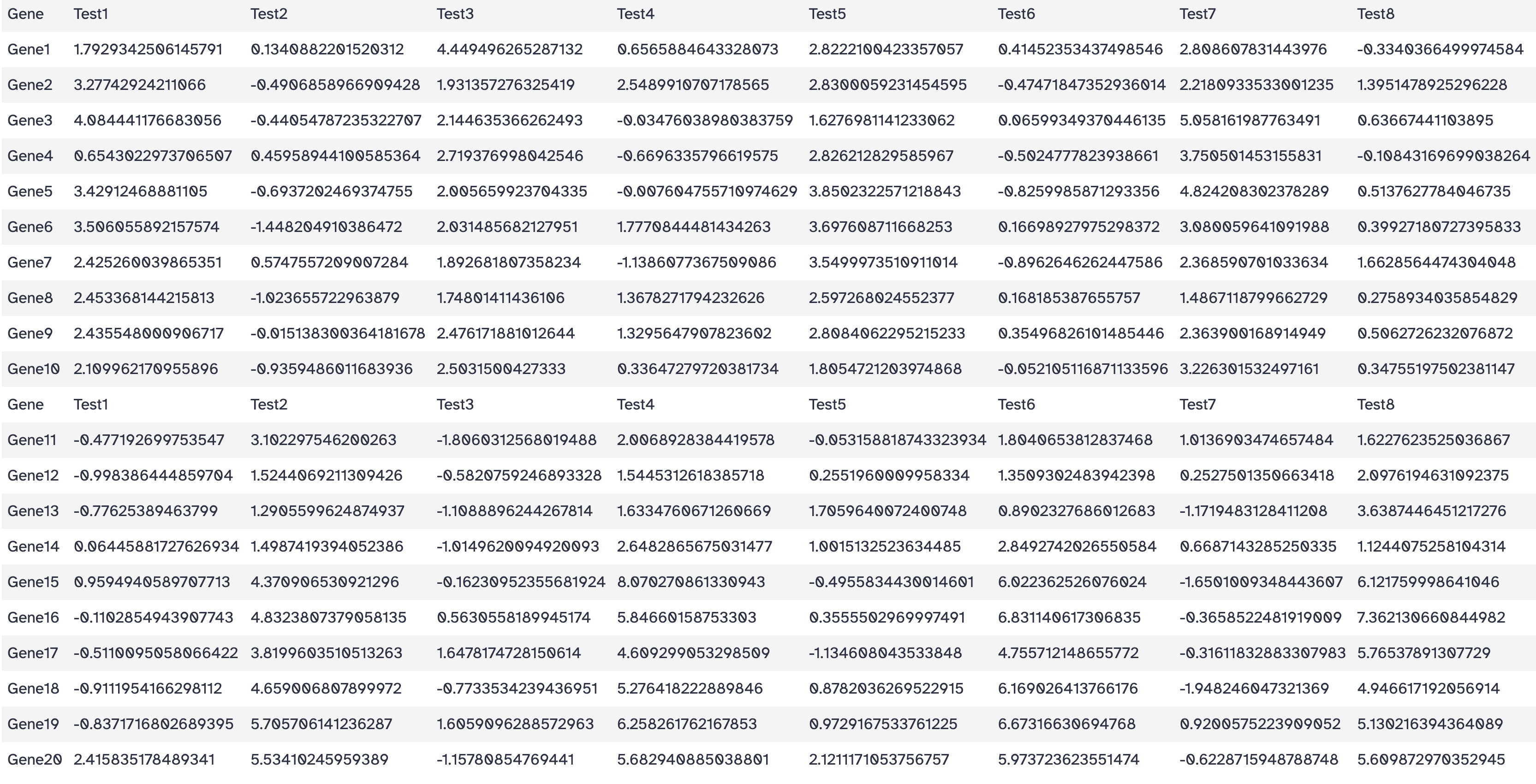
纵向合并多个文件
工具: Text Manipulation > Concatenate datasets tail-to-head
目标: 合并两个文件
操作:
- Concatenate Dataset: Select first on data 1
- Dataset: Select last on data 1
结果:
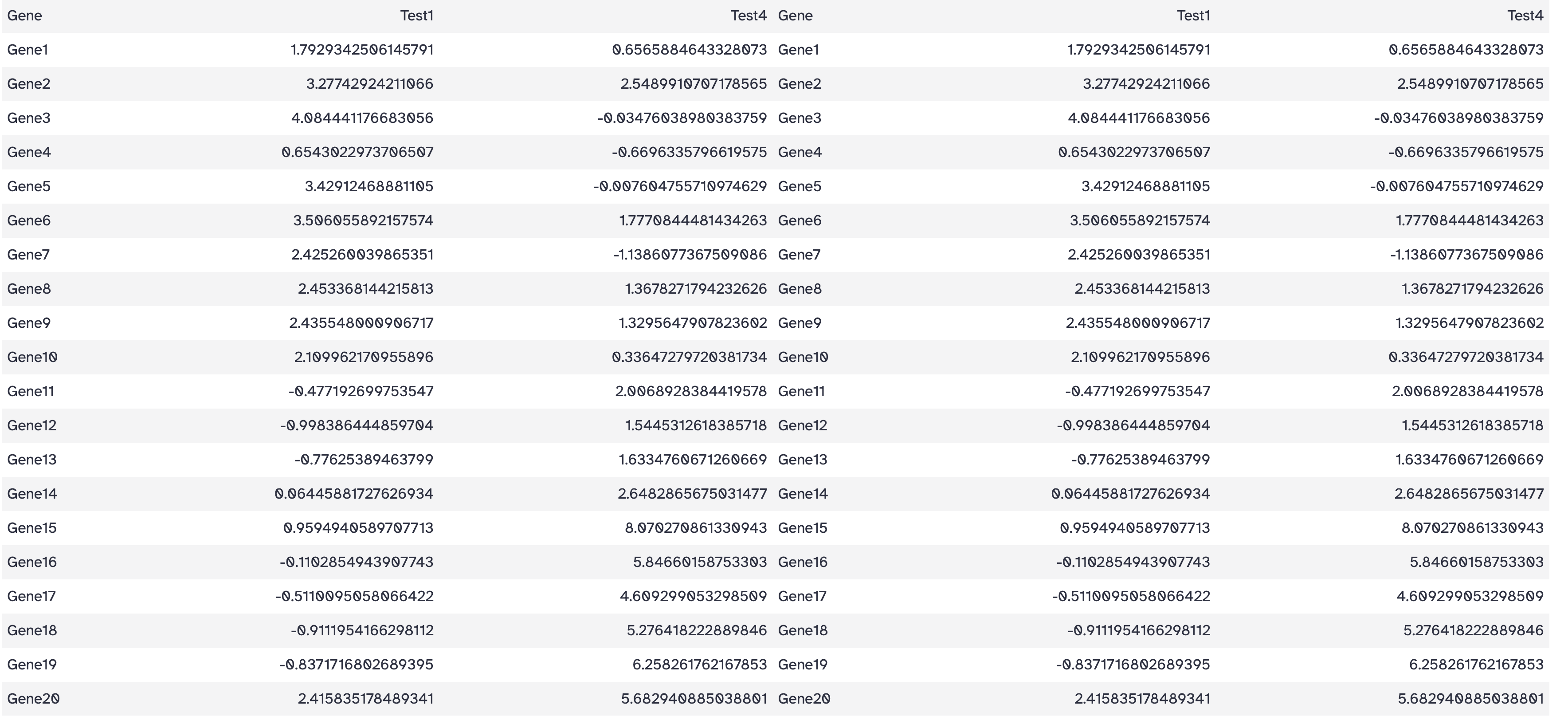
横向合并两个文件
工具: Text Manipulation > Paste two files side by side
目标: 并排粘贴两个文件
操作:
- Paste: Cut on data 1
- and: Cut on data 1
- Delimit by: TAB
结果:
2. 过滤和排序(Filter and Sort)
根据正则表达式匹配行
工具: Filter and Sort > Select lines that match an expression
目标: 筛选出前面纵向合并的文件中的表头
操作:
- Select lines from: Concatenate datasets on data 4 and data 2
- that: Matching
- the pattern: Test1
结果:

目标: 筛选出前面纵向合并的文件中不是表头的行
操作:
- Select lines from: Concatenate datasets on data 4 and data 2
- that: NOT Matching
- the pattern: Test1
结果:

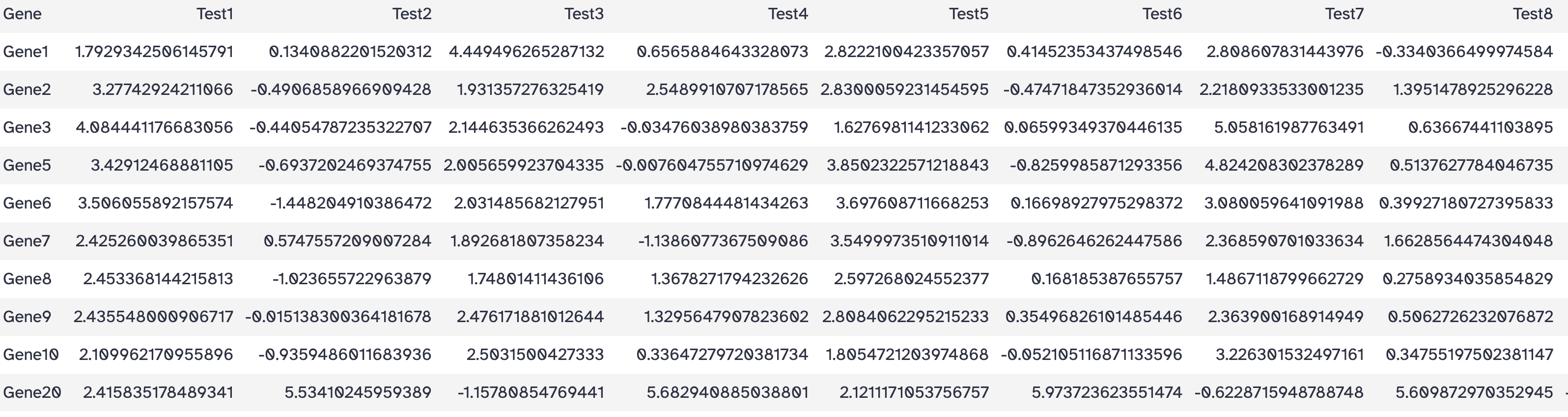
依据某一列过滤数据
工具: Filter and Sort > Filter data on any column using simple expressions
目标: 筛选第2列(即样本Test1所在列)值大于1的行
操作:
- Filter:heatmap_test.tsv
- With following condition: c2 > 1
- Number of header lines to skip: 1
结果:
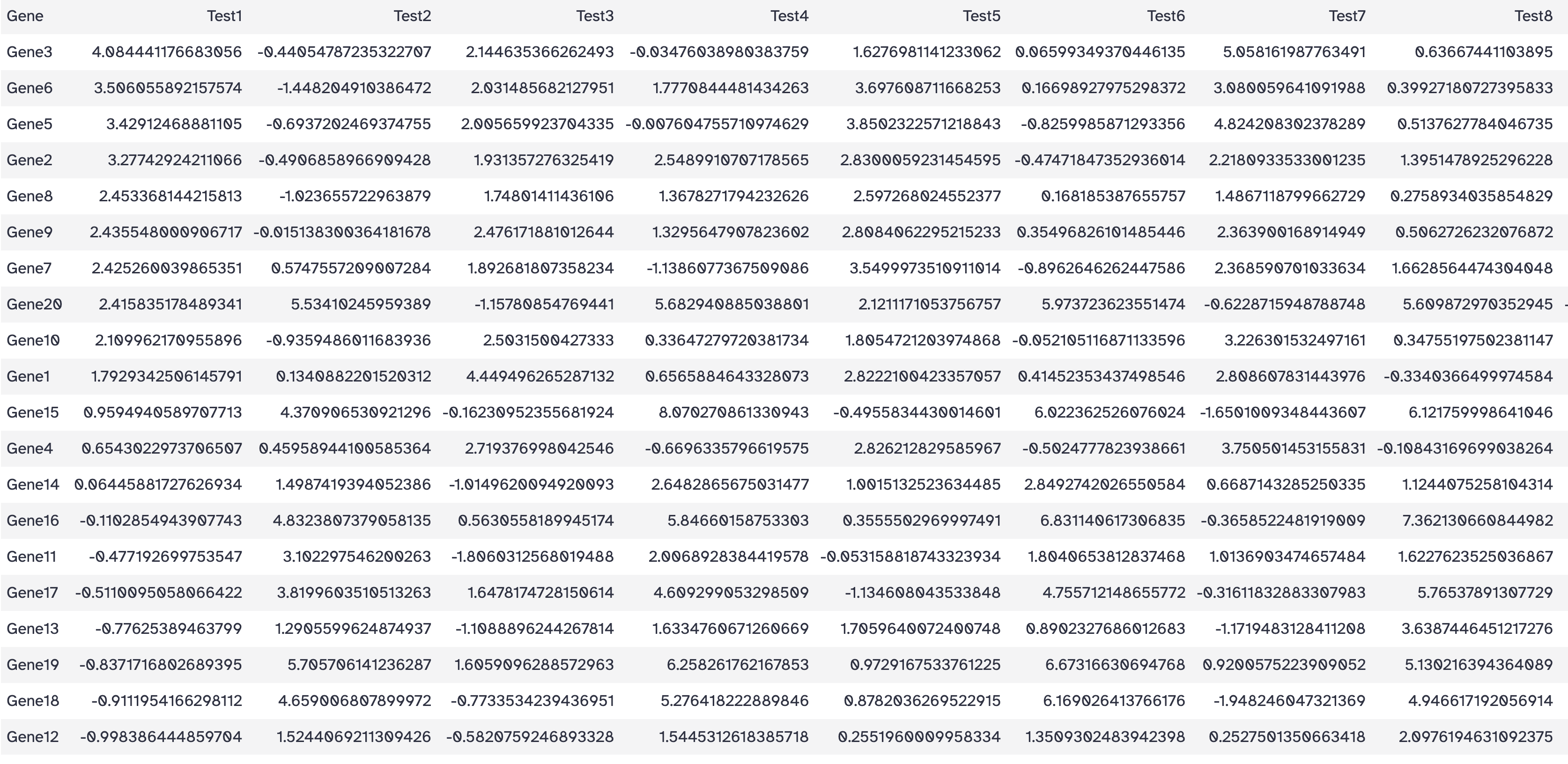
排序数据
工具: Filter and Sort > Sort data in ascending or descending order
目标: 根据表达矩阵的第2列,降序排列行
操作:
- Sort Dataset: heatmap_test.tsv
- on column: Column 2
- with flavor: Numerical sort
- everything in: Descending order
- Number of header lines to skip: 1
结果:
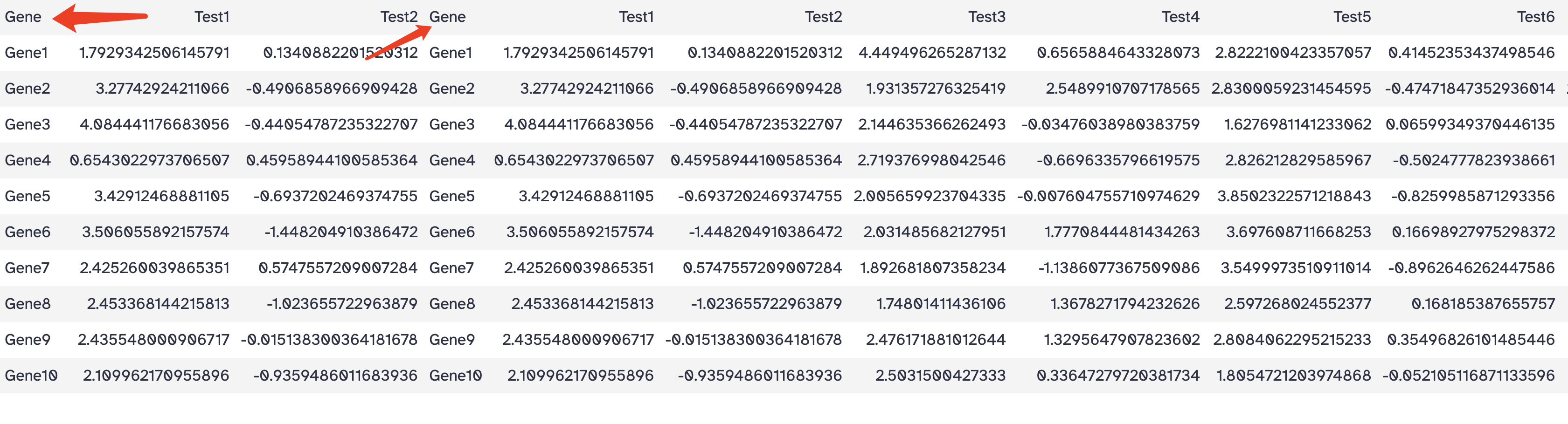
3. 连接、提取和分组(Join, Subtract and Group)
依据特定列连接两个数据表
工具: Text Manipulation > Cut columns
目标: 选择文件前3列
操作:
- Cut columns: c1,c2, c3
- From: Select first on data 1
工具: Join, Subtract and Group > Join two Datasets side by side on a specified field
目标: 连接两个数据表
操作:
- Join: Cut on data 2
- using column: Column: 1
- with: heatmap_test.tsv
- and column: Column: 1
结果:
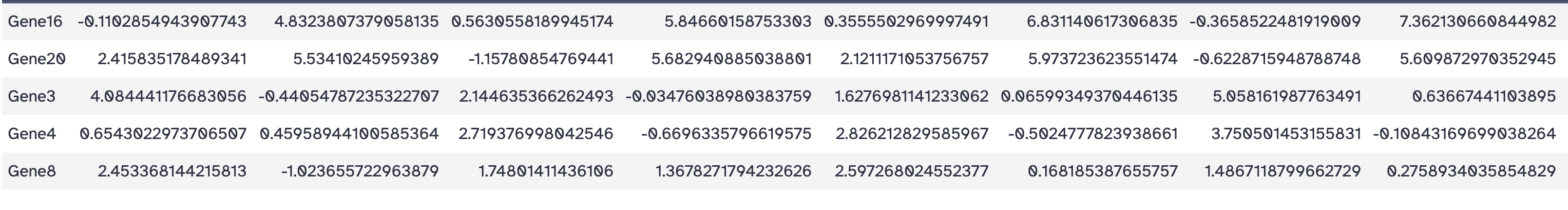
比较两个数据表找出相同或不同的行
工具: Join, Subtract and Group > Compare two Datasets to find common or distinct rows
目标: 查找表1中,某一列的值出现在表2中某一列的行
操作:
- Compare:Select random lines on data 1
- Using column: Column: 1
- against:heatmap_test.tsv
- and column:Column: 1
- To find: Matching rows of 1st dataset
结果:
目标: 查找表1中,某一列的值没有出现在表2中某一列的行
操作:
- Compare:Select random lines on data 1
- Using column: Column: 1
- against:heatmap_test.tsv
- and column:Column: 1
- To find: Non Matching rows of 1st dataset
结果:
因为表1所有的行,都在表2中,因此结果为空。
友情提示
使用问题请加入Galaxy中国交流群: 925694514(QQ群,免费加入),或者关注公众号:简说基因。

